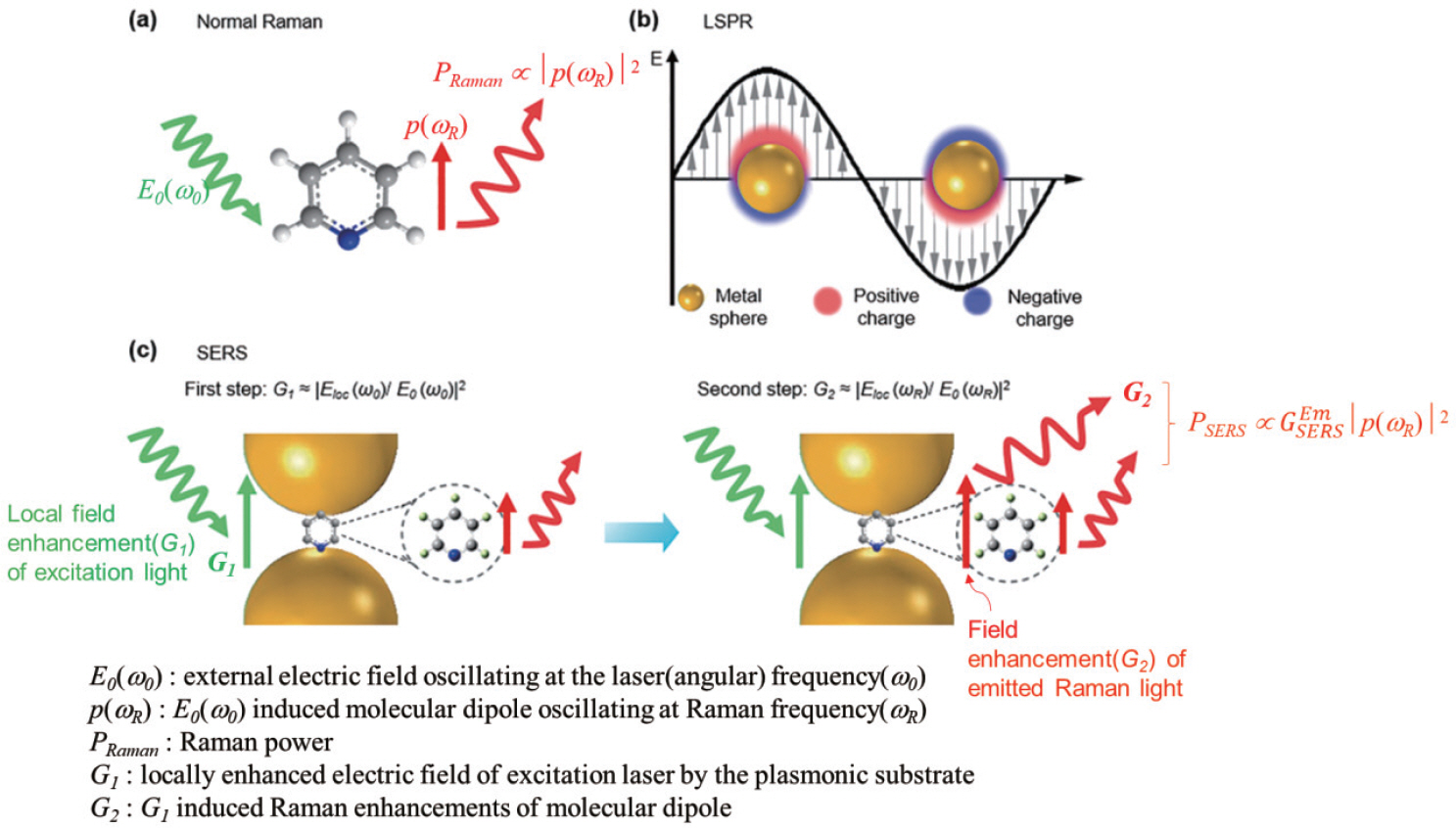
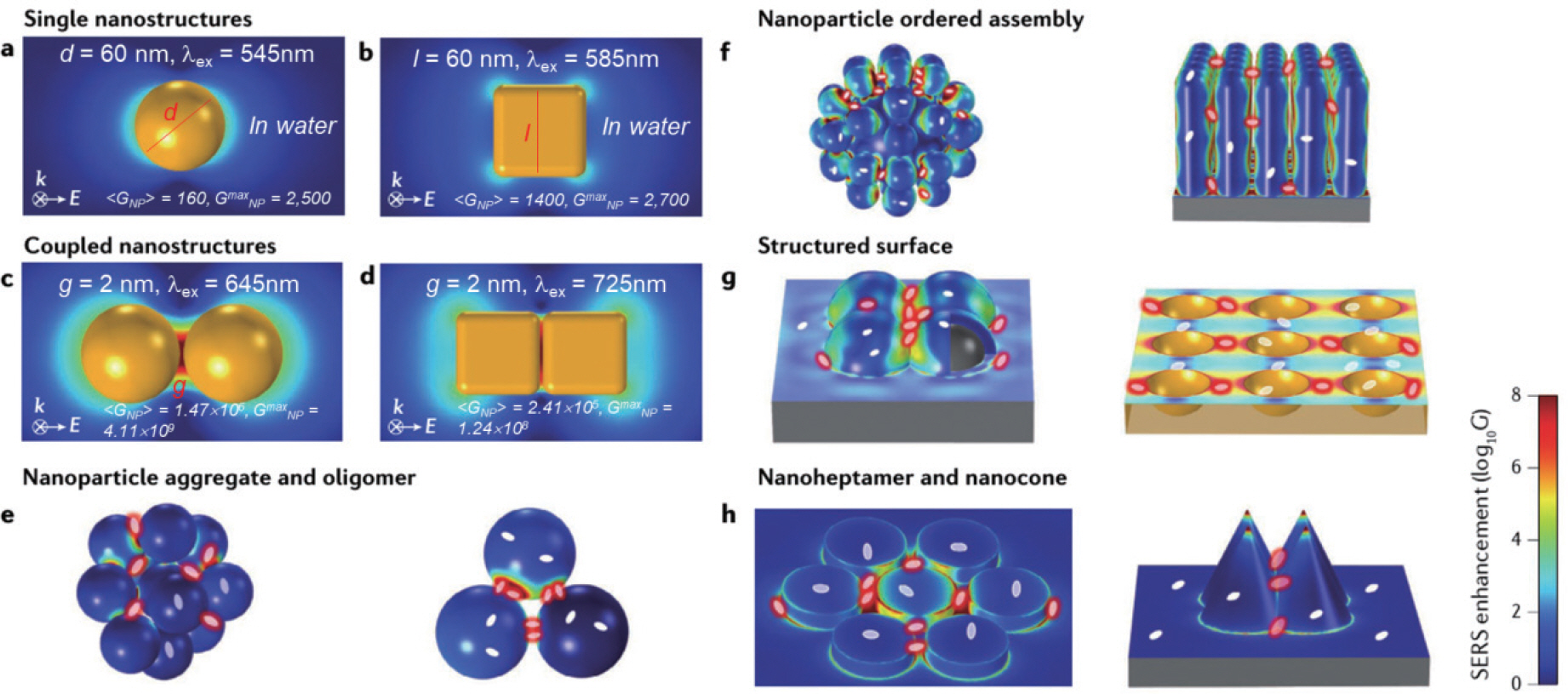
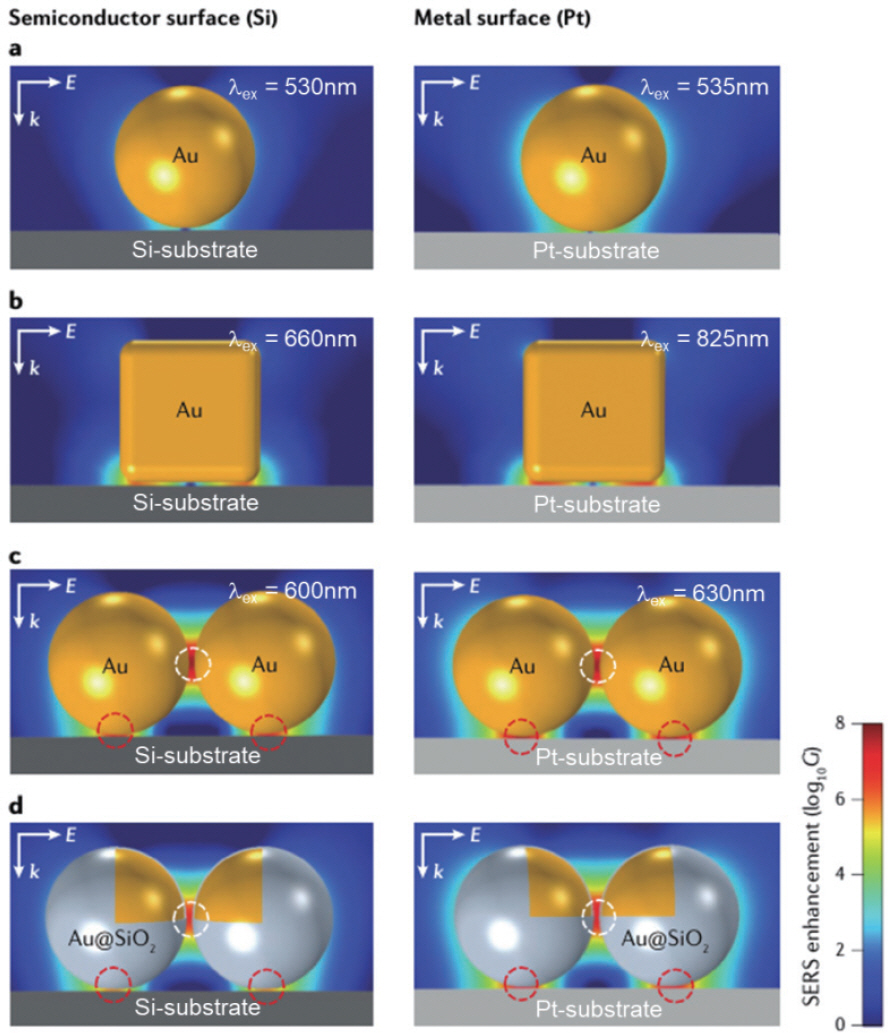
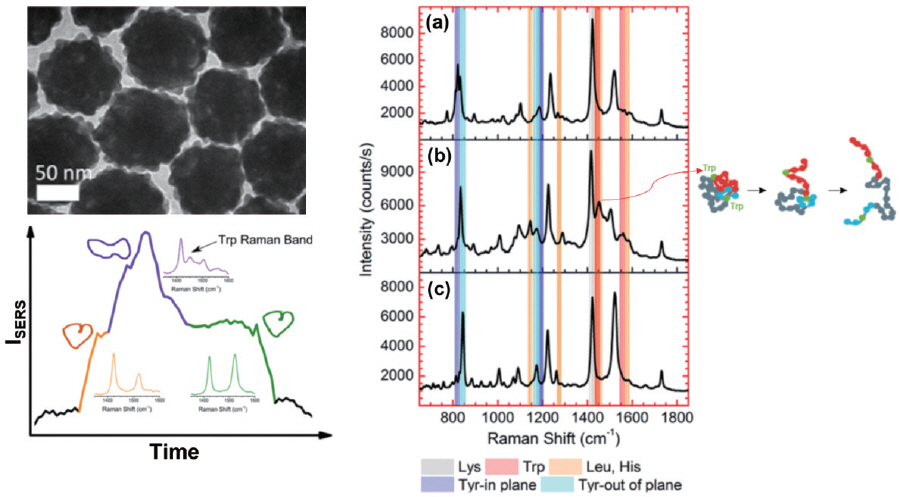
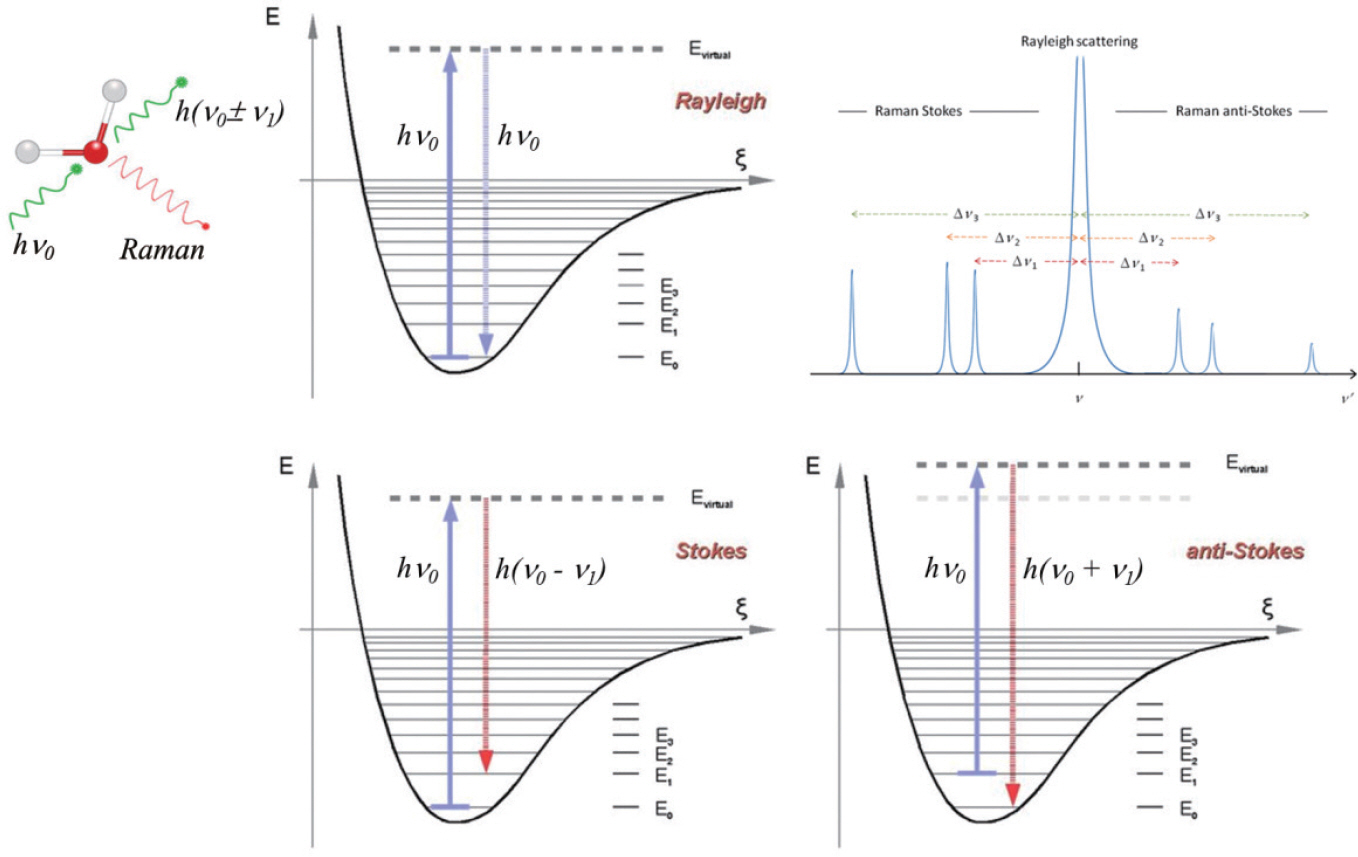
1. M. Fleischmann, P. J. Hendra, A. J. McQuillan, “Raman spectra of pyridine adsorbed at a silver electrode.”
Chem. Phys. Lett..
26(2): 163–166 (1974).

2. M. Moskovits, “Surface roughness and the enhanced intensity of Raman scattering by molecules adsorbed on metals.”
J. Chem. Phys..
69(9): 4159–4161 (1978).

3. M. Moskovits, “Surface-enhanced spectroscopy.”
Rev. Mod. Phys..
57(3): 783–826 (1985).

4. J. A. Creighton, C. G. Blatchford, M. G. Albrecht, “Plasma resonance enhancement of Raman scattering by pyridine adsorbed on silver or gold sol particles of size comparable to the excitation wavelength.”
J. Chem. Soc., Faraday Trans. 2.
75, 790–798 (1979).

5. Y. J. Chen, W. P. Chen, E. Burstein, “Surface-electromagnetic-wave-enhanced Raman scattering by overlayers on metals.”
Phys. Rev. Lett..
36(20): 1207–1210 (1976).

6. R. Dornhaus, R. E. Benner, R. K. Chang, I. Chabay, “Surface plasmon contribution to SERS.”
Surf. Sci..
101(1-3): 367–373 (1980).

7. B. Pettinger, U. Wenning, H. Wetzel, “Surface plasmon enhanced Raman scattering frequency and angular resonance of Raman scattered light from pyridine on Au, Ag and Cu electrodes.”
Surf. Sci..
101(1-3): 409–416 (1980).

8. J. C. Tsang, J. R. Kirtley, T. N. Theis, “Surface plasmon polariton contributions to strokes emission from molecular monolayers on periodic Ag surfaces.”
Solid State Com..
35(9): 667–670 (1980).

9. J. I. Gersten, “The effect of surface roughness on surface enhanced Raman scattering.”
J. Chem. Phys..
72(10): 5779–5780 (1980).

10. J. Gersten, A. Nitzan, “Electromagnetic theory of enhanced Raman scattering by molecules adsorbed on rough surfaces.”
J. Chem. Phys..
73(7): 3023–3037.

11. S. L. McCall, P. M. Platzman, “Raman scattering from chemisorbed molecules at surfaces.”
Phys. Rev. B: Condens. Matter Mater. Phys..
22(4): 1660–1662.

12. M. Kerker,
Acc. Chem. Res.. “Electromagnetic model for surface-enhanced Raman scattering (SERS) on metal colloids.”
17(8): 271–277 (1984).

13. H. Metiu, P. Das, “The electromagnetic theory of surface enhanced spectroscopy.”
Annu. Rev. Phys. Chem..
35, 507–536 (1984).

14. G. C. Schatz, “Theoretical studies of surface enhanced Raman scattering.”
Acc. Chem. Res..
17(10): 370–376 (1984).

15. S.–Y. Ding, E.–M. You, Z.–Q. Tian, M. Moskovits, “Electromagnetic theories of surface-enhanced Raman spectroscopy.”
Chem. Soc. Rev..
46, 4042–4076 (2017).


16. K. Kneipp, Y. Wang, H. Kneipp, L. T. Perelman, I. Itzkan, R. R. Dasari, M. S. Feld, “Single molecule detection using surface enhanced Raman scattering (SERS).”
Phys. Rev. Lett..
78(9): 1667–1670 (1997).

17. S. Nie, S. R. Emory, “Probing single molecules and single nanoparticles by surface-enhanced Raman scattering.”
Science.
275(5303): 1102–1106 (1997).


18. D. S. Grubisha, R. J. Lipert, H.-Y. Park, J. Driskell, M. D. Porter, “Femtomolar detection of prostate-specific antigen: an immunoassay based on surface-enhanced Raman scattering and immunogold labels.”
Anal. Chem..
75(21): 5936–5943 (2003).


19. X. Li, S. Ye, X. Luo, “Sensitive SERS detection of miRNA via enzyme-free DNA machine signal amplification.”
Chem. Com.
52(67): 10269–10272 (2016).

20. C. Jiang, R. Liu, G. Han, Z. Zhang, “A chemically reactive Raman probe for ultrasensitively monitoring and imaging the in vivo generation of femtomolar oxidative species as induced by antitumor drugs in living cells.”
Chem. Com..
49(59): 6647–6649 (2013).

21. G. McNay, D. Eustace, W. E. Smith, K. Faulds, D. Graham, “Surface-enhanced Raman scattering (SERS) and surface-enhanced resonance Raman scattering (SERRS): a review of applications.”
Appl. Spect..
65(8): 825–837 (2011).

22. S. Schluecker, “Surface-enhanced Raman spectroscopy: concepts and chemical applications.”
Angew. Chem., Int. Ed..
53(19): 4756–4795 (2014).

23. R. L. McCreeryRaman spectroscopy for chemical analysis. 157, In: Winefordner J. D., editor, John Wiley & Sons, Inc. Pub. Press; New York, NY, USA: 2000.
24. D. A. LongThe Raman effect : a unified treatment of the theory of Raman scattering by molecules. John Wiley & Sons, Inc. Pub. Press.; New York, NY, USA: 2002.
25. D. Tuschel, “Raman thermometry.” Spectroscopy. 31(12): 8–13 (2016).
26. R. Meier, B. Kip, “Determination of the local temperature at a sample during Raman experiments using stokes and anti-stokes Raman bands.”
Appl. Spect..
44(4): 707–711 (1990).

27. R. Meier, B. Kip, “Determination of the local temperature at a sample during Raman experiments using stokes and anti-stokes Raman bands.”
Appl. Spect..
44(4): 707–711 (2000).

28. R. ArocaSurface-enhanced vibrational spectroscopy. John Wiley & Sons, Inc. Pub. Press.; New York, NY, USA: 2006.
29. R. Pilot, R. Signorini, C. Durante, L. Orian, M. Bhamidipati, L. Fabris, “A review on surface-enhanced Raman scattering.”
Biosensors.
9(57): 1–99 (2019).

30. C. Zong, M. Xu, L.–J. Xu, T. Wei, X. Ma, X.–S. Zheng, R. Hu, B. Ren, “Surface-enhanced Raman spectroscopy for bioanalysis: reliability and challenges.”
Chem. Rev..
118(10): 4946–4980 (2018).


31. R. Pilot, R. Signorini, L. Fabris“Surface-enhanced Raman spectroscopy: principles, substrates, and applications.”; p. 89–164 in
Metal nanoparticles and clusters: Advances in synthesis, properties and applications. In: Deepak F. L., editor, Springer Press; Cham, Switzerland: 2018.

32. P. G. Etchegoin, E. C. Le Ru, “A perspective on single molecule SERS: Current status and future challenges.”
Phys. Chem. Chem. Phys..
10(40): 6079–6089 (2008).


33. K. C. Bantz, A. F. Meyer, N. J. Wittenberg, H. Im, Ö. Kurtuluş, S. H. Lee, N. C. Lindquist, S.-H. Oh, C. L. Haynes, “Recent progress in SERS biosensing.”
Phys. Chem. Chem. Phys..
13(24): 11551–11567 (2011).



34. E. C. Le Ru, C. Galloway, P. G. Etchegoin, “On the connection between optical absorption/extinction and SERS enhancements.”
Phys. Chem. Chem. Phys..
8(26): 3083–3087 (2006).


35. A. Otto, “The “chemical” (electronic) contribution to surface-enhanced Raman scattering.”
J. Raman Spect..
36(6-7): 497–509 (2005).

36. L. A. Dick, A. D. McFarland, C. L. Haynes, R. P. Van Duyne, “Metal film over nanosphere (MFON) electrodes for surface-enhanced Raman spectroscopy (SERS): Improvements in surface nanostructure stability and suppression of irreversible loss.”
J. Phys. Chem. B.
106(4): 853–860 (2002).

37. J. R. Lombardi, R. L. Birke, “A unified view of surface-enhanced Raman scattering.”
Acc. Chem. Res..
42, 734–742 (2009).


38. S.-Y. Ding, J. Yi, J.-F. Li, B. Ren, D.-Y. Wu, R. Panneerselvam, Z.-Q. Tian, “Nanostructure-based plasmon-enhanced Raman spectroscopy for surface analysis of materials.”
Nat. Rev. Mater..
1, 16021(2016).

39. E. C. Le Ru, P. EtchegoinPrinciples of surface enhanced Raman spectroscopy. Elsevier Sci. Press; Amsterdam, The Netherlands: 2009.
40. M. J. Mulvihill, X. Y. Ling, J. Henzie, P. Yang, “Anisotropic etching of silver nanoparticles for plasmonic structures capable of single-particle SERS.”
J. Am. Chem. Soc..
132(1): 268–274 (2010).


41. M. D. Sonntag, J. M. Klingsporn, A. B. Zrimsek, B. Sharma, L. K. Ruvuna, R. P. Van Duyne, “Molecular plasmonics for nanoscale spectroscopy.”
Chem. Soc. Rev..
43(4): 1230–1247 (2014).


42. C. E. Talley, J. B. Jackson, C. Oubre, N. K. Grady, C. W. Hollars, S. M. Lane, T. R. Huser, P. Nordlander, N. J. Halas, “Surface-enhanced Raman scattering from individual au nanoparticles and nanoparticle dimer substrates.”
Nano Lett..
5(8): 1569–1574 (2005).


43. J. M. McLellan, A. Siekkinen, J. Chen, Y. Xia, “Comparison of the surface-enhanced Raman scattering on sharp and truncated silver nanocubes.”
Chem. Phys. Lett..
427(1-3): 122–126 (2006).

44. B. J. Wiley, Y. Chen, J. M. McLellan, Y. Xiong, Z.-Y. Li, D. Ginger, Y. Xia, “Synthesis and optical properties of silver nanobars and nanorice.”
Nano Lett..
7(4): 1032–1036 (2007).


45. J. Fang, S. Liu, Z. Li, “Polyhedral silver mesocages for single particle surface-enhanced Raman scattering-based biosensor.”
Biomaterials.
32(21): 4877–4884 (2011).


46. M. Rycenga, M. H. Kim, P. H. C. Camargo, C. Cobley, Z.–Y. Li, Y. Xia, “Surface-enhanced Raman scattering: Comparison of three different molecules on single-crystal nanocubes and nanospheres of silver.”
J. Phys. Chem. A.
113(16): 3932–3939 (2009).



47. J.-F. Li, Y.-J. Zhang, S.-Y. Ding, R. Panneerselvam, Z.-Q. Tian, “Core–shell nanoparticle-enhanced Raman spectroscopy.”
Chem. Rev..
117(7): 5002–5069 (2017).


48. R. M. Stöckle, Y. D. Suh, V. Deckert, R. Zenobi, “Nanoscale chemical analysis by tip-enhanced Raman spectroscopy.”
Chem. Phys. Lett..
318(1-3): 131–136 (2000).

49. M. S. Anderson, “Locally enhanced Raman spectroscopy with an atomic force microscope.”
Appl. Phys. Lett..
76(21): 3130–3132 (2000).

50. N. Hayazawa, Y. Inouye, Z. Sekkat, S. Kawata, “Metallized tip amplification of near-field Raman scattering.”
Opt. Commun..
183(1-4): 333–336 (2000).

51. B. Pettinger, G. Picardi, R. Schuster, G. Ertl, “Surface enhanced Raman spectroscopy: Towards single molecule spectroscopy.”
Electrochem..
68(12): 942–949 (2000).

52. L. Meng, T. X. Huang, X. Wang, S. Chen, Z. Yang, B. Ren, “Gold-coated AFM tips for tip-enhanced Raman spectroscopy: theoretical calculation and experimental demonstration.”
Optics Express.
23(11): 13804–13813 (2015).


53. N. Kazemi-Zanjani, S. Vedraine, F. Lagugne-Labarthet, “Localized enhancement of electric field in tip-enhanced Raman spectroscopy using radially and linearly polarized light.”
Optics Express.
21(21): 25271–25276 (2013).


54. J. F. Li, Y. F. Huang, Y. Ding, Z. L. Yang, S. B. Li, X. S. Zhou, F. R. Fan, W. Zhang, Z. Y. Zhou, D. Y. Wu, B. Ren, Z. L. Wang, Z. Q. Tian, “Shell-isolated nanoparticle-enhanced Raman spectroscopy.”
Nature.
464(7287): 392–395 (2010).


55. J.-F. Li, S.-Y. Ding, Z.-L. Yang, M.-L. Bai, J. R. Anema, X. Wang, A. Wang, D.-Y. Wu, B. Ren, S.-M. Hou, T. Wandlowski, Z.-Q. Tian, “Extraordinary Enhancement of Raman Scattering from Pyridine on Single Crystal Au and Pt Electrodes by Shell-Isolated Au Nanoparticles.”
J. Am. Chem. Soc..
133(40): 15922–15925 (2011).


56. Y.-F. Huang, C.-Y. Li, I. Broadwell, J.-F. Li, D.-Y. Wu, B. Ren, Z.-Q. Tian, “Shell-isolated nanoparticle-enhanced Raman spectroscopy of pyridine on smooth silver electrodes.”
Electrochim. Acta.
56(28): 10652–10657 (2011).

57. S.-Y. Ding, J. Yi, J.-F. Li, Z.-Q. Tian, “A theoretical and experimental approach to shell-isolated nanoparticle-enhanced Raman spectroscopy of single-crystal electrodes.”
Surf. Sci..
631, 73–80 (2015).

58. D. P. Butcher, S. P. Boulos, C. J. Murphy, R. C. Ambrosio, A. A. Gewirth, “Face-dependent shell-isolated nanoparticle enhanced Raman spectroscopy of 2,2′-bipyridine on Au(100) and Au(111).”
J. Phys. Chem. C.
116(8): 5128–5140 (2012).

59. N. R. Honesty, A. A. Gewirth, “Shell-isolated nanoparticle enhanced Raman spectroscopy (SHINERS) investigation of benzotriazole film formation on Cu(100), Cu(111), and Cu(poly).”
J. Raman Spectrosc..
43(1): 46–50 (2012).

60. A. V. Rudnev, A. Kuzume, Y. Fu, T. Wandlowski, “CO oxidation on Pt(100): New insights based on combined voltammetric, microscopic and spectroscopic experiments.”
Electrochim. Acta.
133, 132–145 (2014).

61. C.-Y. Li, J.-C. Dong, X. Jin, S. Chen, R. Panneerselvam, A. V. Rudnev, Z.-L. Yang, J.-F. Li, T. Wandlowski, Z.-Q. Tian, “In situ monitoring of electrooxidation processes at gold single crystal surfaces using shell-isolated nanoparticle-enhanced Raman spectroscopy.”
J. Am. Chem. Soc..
137(24): 7648–7651 (2015).


62. A. Bonifacio, S. Cervo, V. Sergo, “Label-free surface-enhanced Raman spectroscopy of biofluids: Fundamental aspects and diagnostic applications.”
Anal. Bioanal. Chem..
407(27): 8265–8277 (2015).


63. S. Ma, Q. Li, Y. Yin, J. Yang, D. Liu, “Interference-free surface enhanced Raman scattering tags for single-cell molecular imaging with a high signal-to-background ratio.”
Small.
13(15): 1603340(2017).

64. N. M. S. Sirimuthu, “Investigation of the stability of labelled nanoparticles for SE(R) RS measurements in cells.”
Chem. Com..
47(14): 4099–4101 (2011).

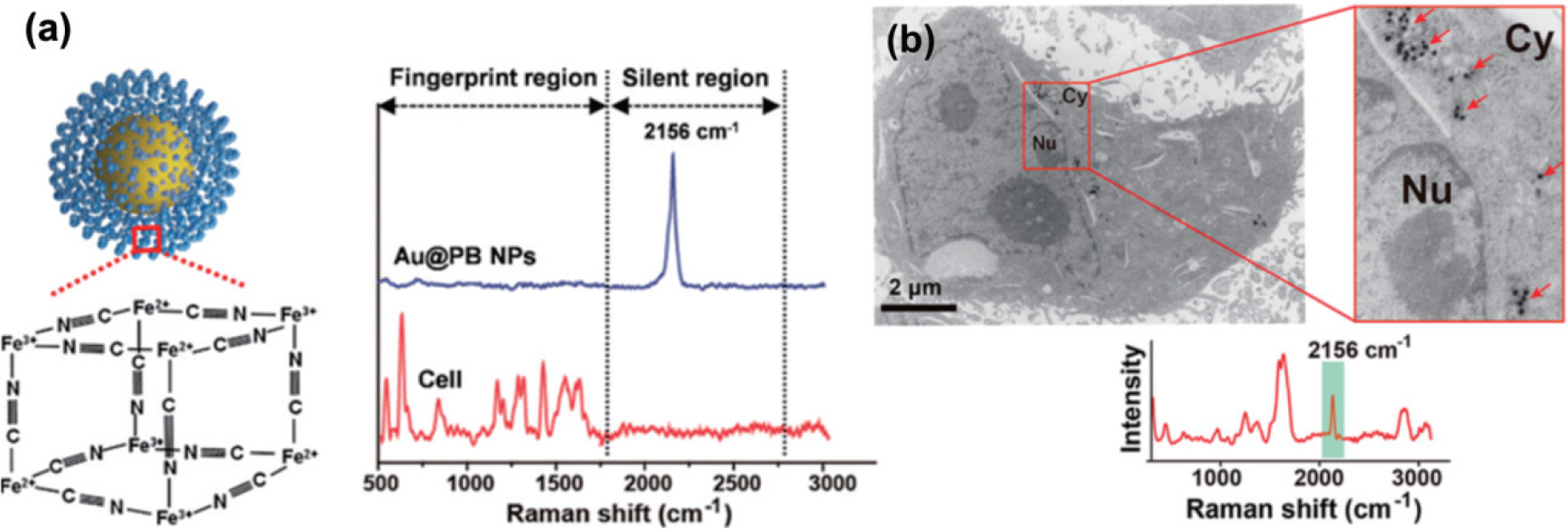
65. Y. Yin, Q. Li, S. Ma, H. Liu, B. Dong, J. Yang, D. Liu, “Prussian blue as a highly sensitive and background-free resonant Raman reporter.”
Anal. Chem..
89, 1551–1557 (2017).


66. K. V. Kong, Z. Lam, W. D. Goh, W. K. Leong, M. Olivo, “Metal carbonyl-gold nanoparticle conjugates for live-cell SERS imaging.”
Angew. Chem., Int. Ed..
51(3): 9796–9799 (2012).

67. Z.-L. Song, Z. Chen, X. Bian, L.-Y. Zhou, D. Ding, H. Liang, Y.-X. Zou, S.-S. Wang, L. Chen, C. Yang, X.-B. Zhang, W. Tan, “Alkyne-functionalized superstable graphitic silver nanoparticles for Raman imaging.”
J. Am. Chem. Soc..
136(39): 13558–13561 (2014).



68. S. Habuchi, J. Hofkens, “Single-molecule surface-enhanced resonance Raman spectroscopy of the enhanced green fluorescent protein EGFP.”
Surf. Enhanced Raman Scattering.
103, 297–312 (2006).

69. K. Singhal, A. K. Kalkan, “Surface-enhanced Raman scattering captures conformational changes of single photoactive yellow protein molecules under photoexcitation.”
J. Am. Chem. Soc..
132(2): 429–431 (2010).


70. H. Xu, E. J. Bjerneld, M. Käll, L. Börjesson, “Spectroscopy of single hemoglobin molecules by surface enhanced Raman scattering.”
Phys. Rev. Lett..
83(21): 4357–4360 (1999).

71. X. X. Han, G. G. Huang, B. Zhao, Y. Ozaki, “Label-free highly sensitive detection of proteins in aqueous solutions using surface-enhanced Raman scattering.”
Anal. Chem..
81(9): 3329–3333 (2009).


72. I. Bruzas, W. Lum, Z. Gorunmez, L. Sagle, “Advances in surface-enhanced Raman spectroscopy (SERS) substrates for lipid and protein characterization: sensing and beyond.”
Analyst.
143(17): 3990–4008 (2018).


73. A. Rygula, K. Majzner, K. M. Marzec, A. Kaczor, M. Pilarczyk, M. Baranska, “Raman spectroscopy of proteins: a review.”
J. Raman Spect..
44(8): 1061–1076 (2013).

74. K. Czamara, K. Majzner, M. Z. Pacia, K. Kochan, A. Kaczor, M. Baranska, “Raman spectroscopy of lipids: a review.”
J. Raman Spect..
46(1):
4–20, (2015).

75. T. Brulé, H. Yockell-Lelièvre, A. Bouhélier, J. Margueritat, L. Markey, A. Leray, A. Dereux, E. Finot, “Spectral pointillism of enhanced Raman scattering for accessing structural and conformational information on single protein.”
J. Phys. Chem. C.
118(1): 17975–17982 (2014).

76. T. Brulé, A. Bouhelier, A. Dereux, E. Finot, “Discrimination between single protein conformations using dynamic SERS.”
ACS Sens..
1(6): 676–680 (2016).

77. L.-J. Xu, Z.-C. Lei, J. Li, C. Zong, C. J. Yang, B. Ren, “Label-free surface-enhanced Raman spectroscopy detection of DNA with single-base sensitivity.”
J. Am. Chem. Soc..
137(15): 5149–5154 (2015).


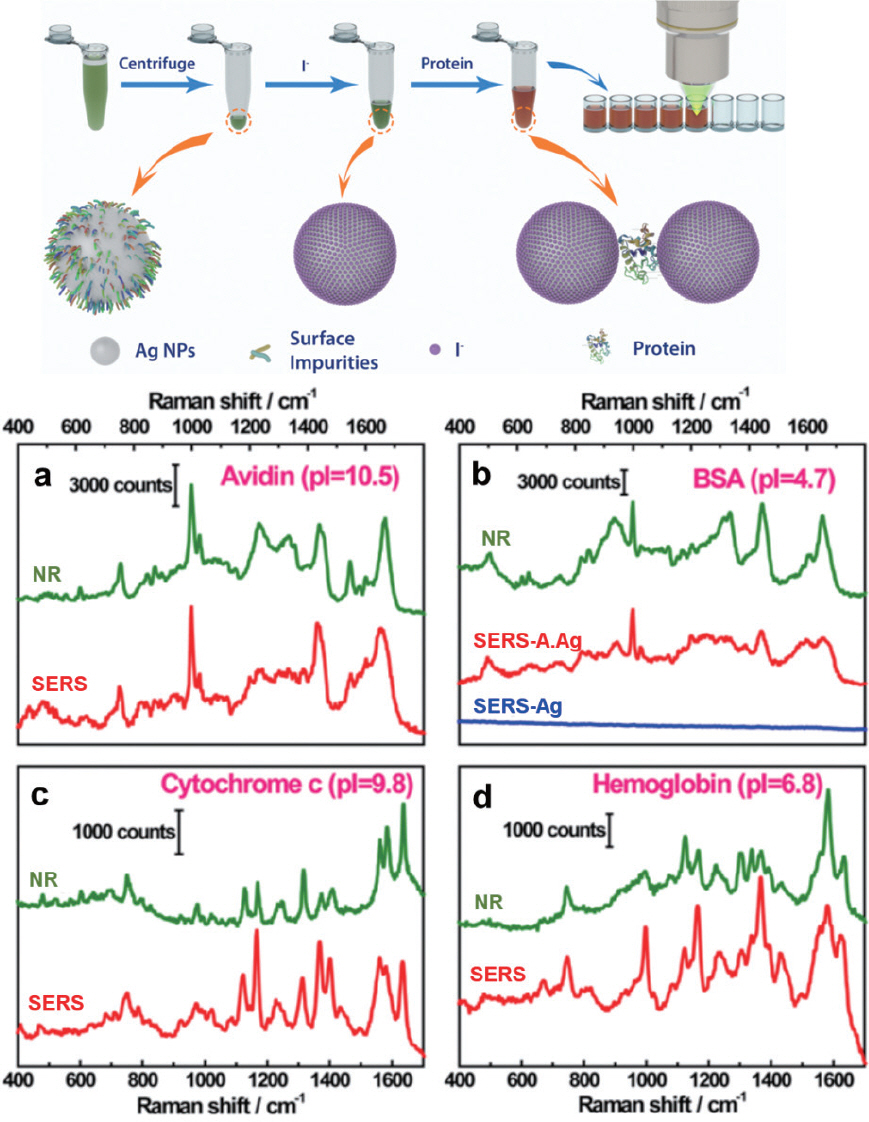
78. L. –J. Xu, C. Zong, X. –S. Zheng, P. Hu, J. –M. Feng, B. Ren, “Label-free detection of native proteins by surface-enhanced Raman spectroscopy using iodide-modified nanoparticles.”
Anal. Chem..
86(4): 2238–2245 (2014).


80. L. Guerrini, Ž. Krpetic´, D. van Lierop, R. A. Alvarez-Puebla, D. Graham, “Direct surface-enhanced Raman scattering analysis of DNA duplexes.”
Angew. Chem., Int. Ed..
54(4): 1144–1148 (2015).

81. E. Garcia-Rico, R. A. Alvarez-Puebla, L. Guerrini, “Direct surface-enhanced Raman scattering (SERS) spectroscopy of nucleic acids: from fundamental studies to real-life applications.”
Chem. Soc. Rev..
47(13): 4909–4923 (2018).


82. E. Papadopoulou, S. E. J. Bell, “Label-free detection of nanomolar unmodified single- and double-stranded DNA by using surface-enhanced Raman spectroscopy on Ag and Au colloids.”
Chem.– Eur. J..
18(17): 5394–5400 (2012).

83. Y. Yin, Q. Li, S. Ma, H. Liu, B. Dong, J. Yang, D. Liu, “Prussian blue as a highly sensitive and background-free resonant Raman reporter.”
Anal. Chem..
89(3): 1551–1557 (2017).


84. Y. Chen, J.-Q. Ren, X.-G. Zhang, D.-Y. Wu, A.-G. Shen, J.-M. Hu, “Alkyne-modulated surface-enhanced Raman scattering-palette for optical interference-free and multiplex cellular imaging.”
Anal. Chem..
88(12): 6115–6119 (2016).


85. D. Radziuk, R. Schuetz, A. Masic, H. Moehwald, “Chemical imaging of live fibroblasts by SERS effective nanofilm.”
Phys. Chem. Chem. Phys..
16(44): 24621–24634 (2014).


86. J. Ando, K. Fujita, N. I. Smith, S. Kawata, “Dynamic SERS imaging of cellular transport pathways with endocytosed gold nanoparticles.”
Nano Lett..
11(12): 5344–5348 (2011).


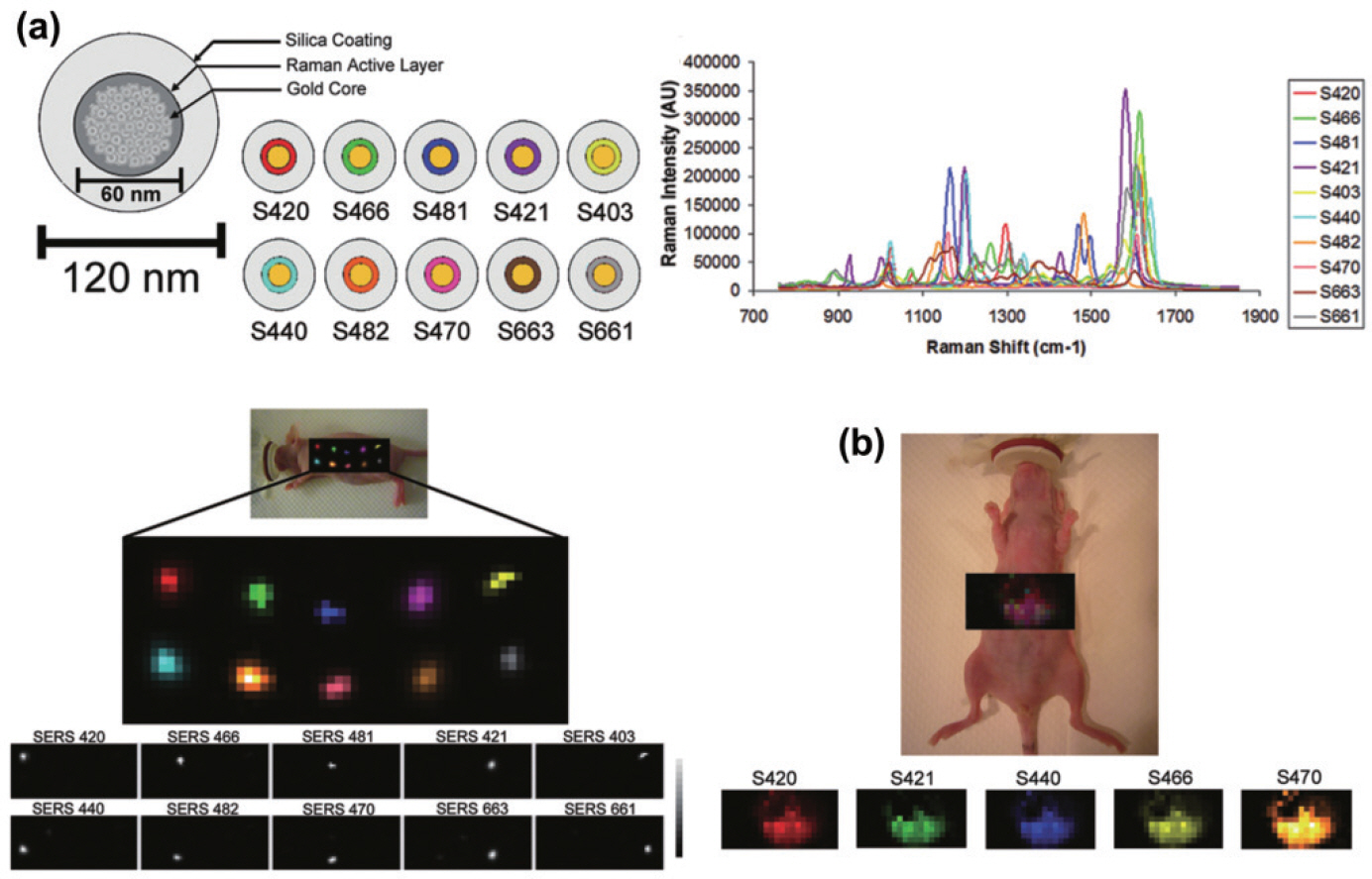
87. C. L. Zavaleta, B. R. Smith, I. Walton, W. Doering, G. Davis, B. Shojaei, M. J. Natan, S. S. Gambhir, “Multiplexed imaging of surface enhanced Raman scattering nanotags in living mice using noninvasive Raman spectroscopy.”
PNAS.
106(32): 13511–13516 (2009).



88. Y. W. Wang, S. Kang, A. Khan, P. Q. Bao, J. T. C. Liu, “In vivo multiplexed molecular imaging of esophageal cancer via spectral endoscopy of topically applied SERS nanoparticles.”
Biomed. Opt. Express.
6(10): 3714–3723 (2015).